EJERCICIO 6
Preparar un conjunto de funciones biotools que permitan automatizar el cálculo de distancias interatómicas, ángulos de enlace y ángulos de torsión a partir de las coordenadas proporcionadas desde un TPDB. Con ayuda de estas herramientas, desarrollar una aplicación Free Pascal/Lazarus que permita construir el diagrama de Ramachandran de la proteína importada. Analizar con este programa algunas de las estructuras de la proteína asignada e incluir el procedimiento y los resultados obtenidos en el CPA. Para confirmar la corrección de los cálculos, incluya también una tabla de ejemplo de los diez primeros residuos, con los valores de torsión calculados y los observados directamente mediante la herramienta “set picking torsión” de RASMOL
Para realizar esta actividad hemos creado una aplicación
denominada ‘Ramachandran’ que nos permitirá observar un diagrama en el que, al
introducir una proteína, nos represente el ángulo fi respecto al psi de los
aminoácidos de esa proteína, donde fi es la rotación del eje que une los átomos
entre Ca y N y psi entre Ca y C. Estos dos enlaces tienen la capacidad de rotar
libremente, por lo que conociendo estos dos ángulos podremos intuir las estructuras
secundarias de las proteínas y diferenciar las zonas hélice alfa (zona más
aglutinada en la parte media del diagrama) y beta hoja plegada (zona más difusa en el
cuadrante superior izquierdo).
Para ello, hemos de ayudarnos de la librería biotools y de
las funciones que hemos ido creando. Para este programa nos ayudamos de la
función ‘distancia’, ‘ángulo’, ‘torsión’ y ‘plotXY’.
A través de la función ‘distancia’ podremos obtener un
número real que se refiere a la distancia entre 2 átomos al introducir como
parámetros de entrada las coordenadas de esos dos puntos. Para que la distancia sea
positiva y debido a que los números enteros a veces causan problemas,
elevaremos las diferencias al cuadrado, las sumaremos y haremos la raíz cuadrada
del resultado.
La función ‘ángulo’ calculará los ángulos de enlace. Para
ello previamente hemos creado las funciones ‘diferenciaV’, ‘móduloV’ y
‘prodEscalar’. En el caso de que los argumentos de entrada sean 2 átomos, la operación
se basa en una sencilla ecuación. En el caso de que me den 3 átomos crearemos
dos vectores diferencia con respecto a uno de los átomos, y usaremos la función
‘ángulo’ con 2 puntos a partir de esas 2 coordenadas.
En la función ‘torsión’ se requieren las coordenadas de los
átomos mediante 4 argumentos de tipo TPunto, y me devolverá un número real
referente a ese ángulo de torsión. Matemáticamente este cálculo equivaldrá a
calcular el ángulo entre 2 planos, ya que el ángulo que forman 2 vectores
perpendiculares al plano será igual al que forman los 2 planos. Esta función
utiliza los cálculos de las funciones ‘diferenciaV’, ‘prodVectorial’, ‘ángulo’,
‘prodEscalar’ y ‘moduloV’ presentes también en biotools.
Para el cálculo de los ángulos, en biotools hemos indicado dentro de la función 'CargarPDB' mediante un bucle los residuos que se han de ir cogiendo para realizar las torsiones necesarias:
Finalmente, la función ‘plotXY’ nos permitirá la representación
de la matriz bidimensional del diagrama. Son necesarios numerosos argumentos,
entre los que destacamos una matriz de tipo TTablaDatos con los resultados de
los ángulos y un parámetro tipo TImage para plasmar la gráfica.
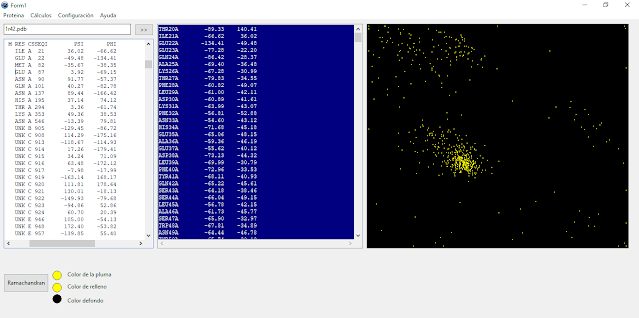
A continuación en la Fig. 1 muestro el resultado al introducir mi
proteína asignada ACE2 en el programa ‘Ramachandran’:
En primer lugar destacamos la gran precisión del programa, ya que los ángulos mostrados en el PDB de la proteína coinciden exactamente con los calculados mostrados en el TMemo con color azul de fondo en la Fig. 1.
Por otro lado, ara el análisis de los resultados con la proteína ACE2 tendremos en cuenta la figura mostrada en la Fig. 2. De esta forma, observamos que mi proteína tiene una nube difusa en el cuadrante superior izquierdo, que hace referencia a las estructuras beta hoja plegada. Por otro lado, la nube más densa del centro es debido a hélices alfa. Cabe destacar que las hélices alfa son mucho más abundantes que las hojas beta, lo cual es coherente con la bibliografía, ya que ACE2 es una proteína transmembrana y, por tanto, tendrá muchas regiones alfa hélice embebidas en la membrana plasmática.

Fig 2. Relación de ángulos fi y psi en el gráfico Ramachandran.
Por otro lado, otra característica del programa es que me permite, mediante la elección de plumas de distintos colores, comparar entre distintas estructuras proteicas.
A continuación, en la Fig. 3. se muestra la comparación entre IR42.pdb (amarillo) y 6LZG.pdb (azul), dos estructuras de la proteína ACE2:
Fig. 3. Diagrama ramachandran para las estructuras IR42.pdb (color amarillo) y 6LZG.pdb (color azul) de la proteína asignada ACE2.
El resultado esperado era que ambas estructuras tuvieran una altísima semejanza en su estructura, ya que se refieren a la misma proteína y, como sabemos, la estructura y la función de la proteína se encuentran íntimamente relacionadas. Los observado en la Fig. 3. es que las nubes de puntos se encuentran en las mismas zonas, aunque con pequeñas diferencias. 6LZG.pdb también muestra numerosas hélices alfa y diversas hojas beta, además de compartir otras estructuras como las hélices alfa levógiras que se observan en la zona central derecha.
A continuación, gracias a la página web Molprobity, voy a comparar los ángulos de torsión mostrados en la Fig. 1 con los que me devuelva este programa:
Fig. 4. Ángulos de torsión devueltos por Molprobity al introducir la estructura 1r42.pdb de la proteína ACE2.
Comparando la Fig. 4. con la Fig. 1. observamos que los datos son exactos. Si existiesen pequeñas variaciones en alguno de los residuos se debería probablemente a los decimales que se tienen en cuenta al calcular los ángulos fi y psi.




Comentarios
Publicar un comentario