EJERCICIO 11
Elaborar una aplicación informática que permita predecir (“asignar”) los posibles enlaces disulfuro presentes en una proteína a partir de sus datos estructurales contenidos en el PDB. Realizar un breve muestreo con media docena de proteínas y comentar los resultados obtenidos en términos de aciertos positivos, aciertos negativos, falsos positivos y falsos negativos.
Los enlaces disulfuro son uniones covalentes que se producen
mayoritariamente entre dos grupos tiol y, en el caso de las proteínas, ocurren
entre los azufres de las cisteínas y son esenciales para el plegamiento e
integridad de la estructura terciaria y para la regulación de numerosos
procesos.
El criterio que estableceremos para suponer si se forma
enlace disulfuro entre 2 Cys es que estén a una determinada distancia. Sin
embargo, en la práctica van a intervenir otros factores, como por ejemplo el
ambiente óxido-reductor en que se encuentre la proteína, ya que las cisteínas
pueden encontrarse protonadas y no formar enlace. Por ello, es posible que el
programa que creemos de falsos positivos (nos muestra un enlace que no existe
en la realidad) o falsos negativos (ignora un enlace que sí que hay en la
práctica).
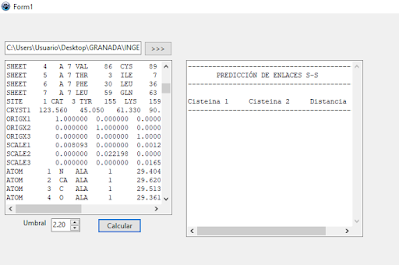
Crearemos una aplicación denominada ‘pruebaSS’, en el que
recogeremos la distancia que establecemos como criterio de enlace en una
variable ‘umbral’. En el formato PDB el ID de las Cys es ‘SG’, que es lo que
usaremos para buscarlas al recorrer el Memo en el que se encuentra la proteína IR42.pdb. Una vez calculadas las distancias, las introducimos en
una matriz distancia y, aquellas que entren en el umbral establecido, se
plasmarán en una tabla.
Para todo ello, dentro del Button2 anido 3 procedimientos
que me realicen lo explicado (buscar_SG, calcular_dist, mostrar_resultado).
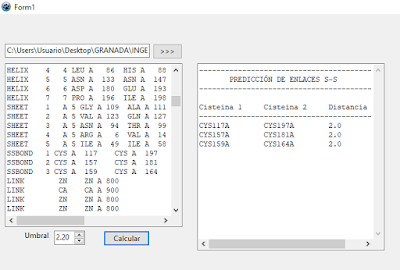
Al introducir mi proteína ACE2, estableciendo una distancia
de 2.2A como criterio, obtengo enlaces entre Cys133-Cys141, Cys 344-Cys361 y Cys530-Cys542:
Fig. 1. Resultado mostrado al ejecutar la aplicación 'pruebaSS' con IR42.pdb.
Comparando con la bibliografía del Ejercicio 1 vemos que los resultados son los mismos a los que predice la aplicación, por lo que podemos concluir una alta fiabilidad de la aplicación creada.
A continuación, introduciré en la aplicación una serie de proteínas ejemplo para ver los puentes predichos y estudiar la fiabilidad del programa más a fondo. La distancia umbral en todos los casos será de 2.2A, ya que considero que es una distancia suficientemente grande como para omitir ciertos enlaces.
Fig. 2. Puentes disulfuro hallados en 1AFD.pdb
Fig. 3. Puentes disulfuro hallados en 1AHI.pdb
Fig. 4. Puentes disulfuro hallados en 1BHS.pdb
Fig. 5. Puentes disulfuro hallados en 1BUD.pdb
Fig. 6. Puentes disulfuro hallados en 9WGA.pdb
Fig. 7. Puentes disulfuro hallados en 12GS.pdb






Comentarios
Publicar un comentario