EJERCICIO 2
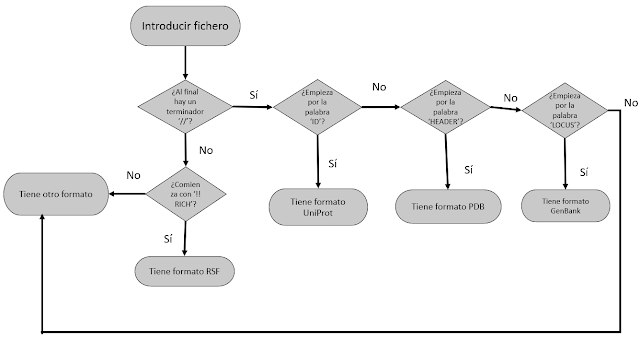
Desarrollar un diagrama de flujo que permita identificar si un fichero tiene formato EMBL, UniProt, GenBank, PDB u otros diferentes. A continuación, desarrollar un programa informático capaz de discriminar automáticamente entre tres de ellos: Genbank y UniProt y PDB y muestre en pantalla las secuencias primarias de las proteínas correspondientes en códigos de una letra. Emplear ese programa con los ficheros bajados de Internet de la proteína asignada. Sugerencia: Estudiar detenidamente la página del EBI dedicada a bases de datos (se incluye una copia con información útil al respecto en el CD con el nombre “formatos de secuencias y estructuras.pdf”).
Para este ejercicio he creado una aplicación denominada ‘Formatos’
en la que abriremos nuestra proteína para leerla en un TMemo y recorreremos la
primera línea de ese memo para determinar el tipo de formato que tiene,
diferenciando entre el formato PDB (precedido por ‘HEADER’), formato UniProt
(precedido por ‘ID’) y formato GenBank (precedido por ‘LOCUS’).
Una vez sepamos el formato, volveremos a recorrer el memo
buscando su secuencia de aminoácidos en código de una letra, que lo pondremos a continuación en otro TMemo. En el caso de que sea formato PDB usaremos la función de biotools
‘AA3to1’ que convierte la secuencia de aminoácidos de código de 3 letras (tipo string) en un
código de una letra (tipo char).
En el caso de que el formato sea GenBank, el programa recorrerá
las líneas del memo hasta encontrar ‘/translation=’, puesto que es lo que
precede a la secuencia de aminoácidos, y empezará a copiar a partir de ahí,
deteniéndose donde se encuentre dos comillas (“), que significará el fin de la
secuencia (ver Fig. 2.).
En caso de que la primera palabra sea ‘ID’ y tenga formato UniProt, deberemos buscar entonces las letras ‘SQ’, que indican el comienzo de la secuencia, y copiar todo hasta el final (ver Fig. 3.).
En tercer lugar, en caso de que el formato sea PDB,
deberemos buscar las líneas precedidas por ‘SEQRES’, ya que en ellas se
encuentra la secuencia de aminoácidos en código de 3 letras. Una vez hecho
esto, como se ha explicado, llamaremos a la función 'AA3to1' que, en caso de no
reconocer el aminoácido introducido, nos devolverá un asterisco (*) que no
añadiremos al memo que contiene la secuencia
A continuación mostraré lo que devuelve mi aplicación al abrir archivos con cada uno de los formatos:





Comentarios
Publicar un comentario